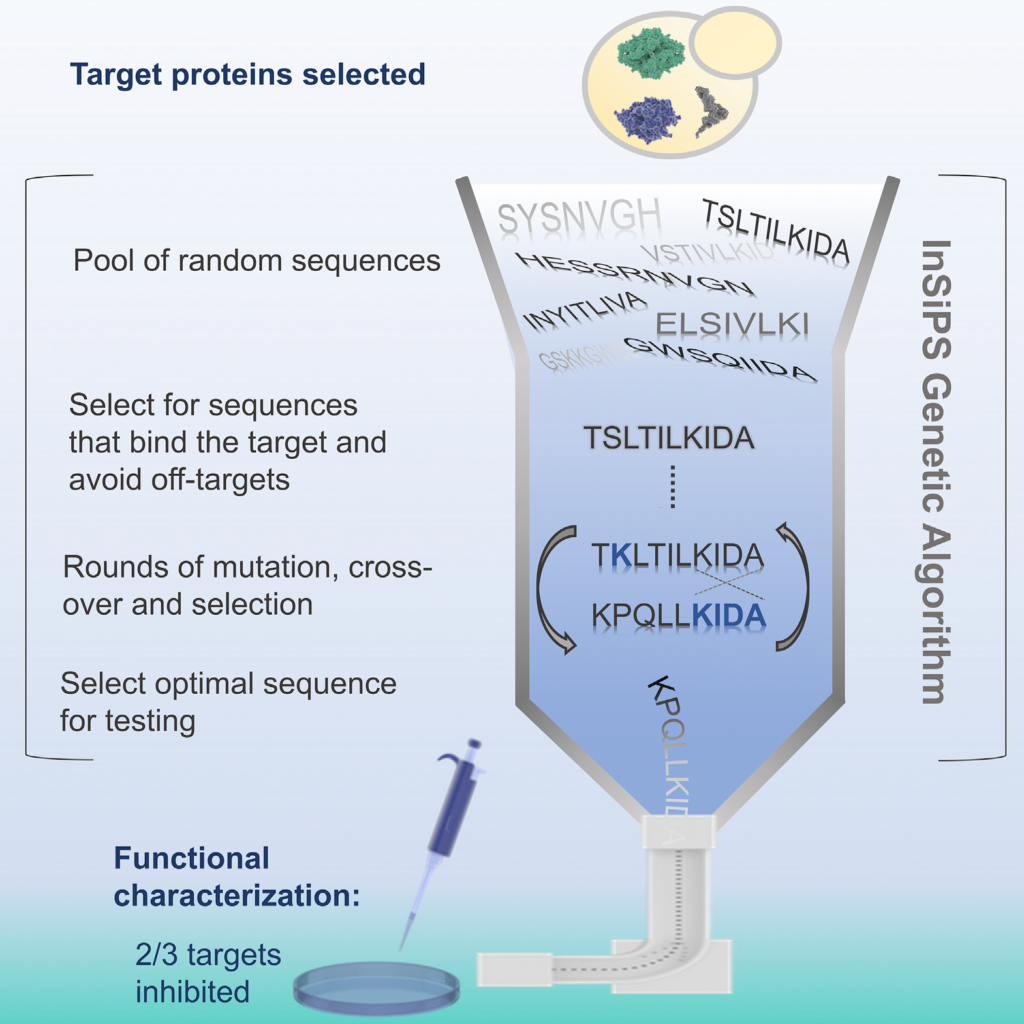
Syntetyczne białka wykazujące wysokie powinowactwo i selektywność dla białek docelowych mogą być wykorzystywane jako narzędzia badawcze, biomarkery i czynniki farmakologiczne, istnieje jednak niewiele metod pozwalających na projektowanie takich białek de novo. W tym celu stworzony został system syntezy białek „in silico” tzw. In-Silico Protein Synthesizer (InSiPS) który służy projektowaniu syntetycznych białek wiążących (synthetic binding proteins SBP), które łączą się pre – definiowanymi grupami docelowymi, jednocześnie minimalizując niespecyficzne interakcje z innymi białkami. InSiPS jest algorytmem genetycznym, który umożliwia wyselekcjonowanie puli losowych sekwencji, pośród setek różnych generacji mutacji, w celu wytworzenia SBP z wcześniej określoną charakterystyką wiązania. Jako potwierdzenie koncepcji, autorzy zaprojektowali SBP przeciwko trzem białkom drożdży i wykazali możliwość wiązanie tych białek i ich funkcjonalnego hamowania in vivo. Tego rodzaju podejście może istotnie wspierać dziedzinę projektowania de novo małych łańcuchów wiążących polipeptydy, może mieć istotne znaczenie praktyczne, i być bardzo przydatne szczególnie wobec ograniczonej liczby obecnie dostępnych technik projektowania białek „in silico”. ( Daniel Burnside, Andrew Schoenrock, Houman Moteshareie, Mohsen Hooshyar, Prabh Basra, Maryam Hajikarimlou, Kevin Dick, Brad Barnes, Tom Kazmirchuk, Matthew Jessulat, Sylvain Pitre, Bahram Samanfar, Mohan Babu, James R. Green, Alex Wong, Frank Dehne, Kyle K. Biggar, and Ashkan Golshan. In Silico Engineering of Synthetic Binding Proteins from Random Amino Acid Sequences. iScience 11, 375–387, January 25, 2019 .).
https://doi.org/10.1016/j.isci.2018.11.038